Herramientas bioinformáticas para mejorar el diagnóstico de virus y el seguimiento de epidemias
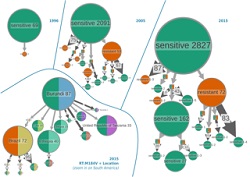
Los avances recientes en las tecnologías de secuenciación incluyen el desarrollo de la SNG y de análisis genéticos rápidos mediante bioinformática de individuos y poblaciones. La SNG se está convirtiendo en una práctica rutinaria en los laboratorios científicos y clínicos de los sectores público y privado. Sin embargo, a pesar de que se dispone de millones de fragmentos de secuencias generadas por plataformas de SNG, hasta hace poco solo se utilizaba una pequeña parte para el descubrimiento de virus. El principal escollo es que los métodos bioinformáticos de vanguardia actuales tardan mucho tiempo en clasificar y ordenar con rapidez y precisión virus conocidos y desconocidos utilizando datos metagenómicos de SNG disponibles. El objetivo principal del consorcio VIROGENESIS era desarrollar nuevos métodos computacionales y «software» para superar las limitaciones bioinformáticas en el análisis y la traducción de los resultados de SNG para aplicaciones clínicas y de salud pública en el campo de la virología. Otro objetivo importante fue el desarrollo de nuevos métodos para caracterizar cambios en el viroma al instante durante epidemias víricas utilizando grandes conjuntos de datos de SNG de virus. Por último, los socios del proyecto querían proporcionar un «software» fácil de usar que permitiera una visualización dinámica e interactiva de brotes epidémicos casi al instante. Herramientas bioinformáticas para el descubrimiento de virus y el análisis de datos clínicos El consorcio VIROGENESIS logró desarrollar algoritmos y aplicaciones de «software» para el análisis de datos de SNG que solventaban los principales escollos en la investigación de la clasificación y la ordenación de virus. Este impresionante desarrollo desembocó en la creación de Genome Detective, un marco bioinformático basado en la web completamente funcional para la identificación rápida de todas las familias de virus conocidas utilizando datos de SNG y secuenciación Sanger. El aumento de la flexibilidad y la velocidad de las nuevas aplicaciones permite el análisis rutinario de cuestiones relacionadas con el diagnóstico, la filogeografía, la filodinámica y la transmisión de farmacorresistencia. Las herramientas de VIROGENESIS aceleran la traducción de los resultados de los análisis de SNG a la investigación de cambios microbianos, la fuente de transmisión, el origen geográfico y la extensión de la propagación de patógenos. Es más, ya se han hecho públicos varios desarrollos finales del proyecto para análisis genómicos. Entre ellos se incluye UGENE, un «software» de escritorio, multiplataforma y de código abierto que incorpora un conjunto de herramientas bioinformáticas ampliamente utilizadas. VIROGENESIS colaboró asimismo con LUCIAD, la empresa que desarrolla «software» para soluciones geoespaciales de conciencia situacional. Esta colaboración está ayudando a crear una plataforma para la visualización dinámica y la identificación de factores ecológicos, demográficos y epidemiológicos que influyen en la propagación de virus. Aplicación de VIROGENESIS en los brotes víricos actuales Durante la reciente brote epidémico del virus del Ébola, se desplegaron varios equipos internacionales de investigación para secuenciar este virus con fines de investigación y de salud pública. El «software» de VIROGENESIS demostró ser muy eficaz en varios estudios relacionados con brotes recientes en África y Sudamérica. El marco de descubrimiento Genome Detective identifica patógenos víricos directamente a partir de datos de SNG. El uso de estos datos junto con las herramientas adicionales de VIROGENESIS proporciona visualizaciones informativas e interfaces gráficas de usuario dinámicas que permiten el seguimiento espacial y temporal de epidemias. A través de un estudio se presentó un ejemplo de detección eficaz de patógenos víricos. En él se utilizaron las herramientas de VIROGENESIS y datos de secuenciación en tiempo real para el seguimiento genómico, espacial y epidemiológico de la epidemia del virus de la fiebre amarilla de 2018 en Brasil. VIROGENESIS permite el análisis eficiente de datos de SNG para el descubrimiento, el diagnóstico y el seguimiento de virus. Tal y como manifiestan los coordinadores del proyecto: «Con el aumento de la obtención de datos genómicos de SNG relativos a epidemias y brotes emergentes de virus, prevemos un mayor uso de las herramientas de VIROGENESIS para el análisis de estos datos, donde se requiere una rápida identificación del tipo de virus y de la fuente de transmisión».
Palabras clave
VIROGENESIS, SNG, bioinformática, «software», brotes víricos, descubrimiento de virus