Narzędzia bioinformatyczne posłużą do rozwoju diagnostyki wirusów i monitorowania epidemii
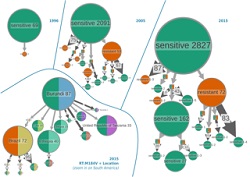
Najnowsze osiągnięcia w pracach nad technologiami sekwencjonowania obejmują opracowanie opartych na NGS i bioinformatyce błyskawicznych analiz genetycznych poszczególnych osób i całych populacji. Analiza NGS staje się rutynową procedurą wykonywaną w laboratoriach badawczych i klinicznych zarówno w sektorze publicznym, jak i prywatnym. Jednakże pomimo dostępności milionów fragmentów sekwencji generowanych przez platformy NGS jeszcze do niedawna do wykrywania wirusów wykorzystywano zaledwie ich ułamek. Główną przeszkodą było to, że obecne metody stosowane w bioinformatyce są zbyt powolne, by szybko i dokładnie sklasyfikować i zgromadzić znane i nieznane wirusy przy użyciu dostępnych danych NGS z metagenomów. Głównym celem konsorcjum VIROGENESIS było opracowanie nowych metod obliczeniowych oraz oprogramowania, które umożliwią pokonanie przeszkód bioinformatycznych stojących na drodze do analizy i translacji wyników NGS na potrzeby zastosowań klinicznych i związanych ze zdrowiem publicznym w dziedzinie wirusologii. Innym ważnym celem było opracowanie nowatorskich metod charakteryzacji zmian wiromu w czasie rzeczywistym w trakcie epidemii wirusowych z wykorzystaniem dużych zestawów danych NGS na temat wirusów. Ostatnim celem partnerów projektu było opracowanie przyjaznego dla użytkownika oprogramowania umożliwiającego dynamiczną i interaktywną wizualizację wybuchu epidemii niemal w czasie rzeczywistym. Narzędzia bioinformatyczne służące do wykrywania wirusów i analizy danych klinicznych Konsorcjum VIROGENESIS z powodzeniem opracowało algorytmy i oprogramowanie służące do analizy danych NGS, pokonując główne przeszkody utrudniające badania nad gromadzeniem i klasyfikacją wirusów. Te imponujące wyniki skutkowały utworzeniem w pełni funkcjonalnej internetowej sieci bioinformatycznej Genome Detective, umożliwiającej szybką identyfikację wszystkich znanych rodzin wirusów z wykorzystaniem danych sekwencjonowania NGS i Sanger. Większa elastyczność i szybkość nowych aplikacji umożliwiają codzienną analizę zagadnień takich jak diagnostyka, filogeografia, filodynamika oraz przenoszenie odporności na leki. Narzędzia opracowane przez VIROGENESIS przyspieszają przekształcanie wyników analizy NGS w badania nad zmianami drobnoustrojów, źródłem zakażenia, pochodzeniem geograficznym oraz zakresem rozprzestrzeniania się patogenów. Co ważne, udostępniono kilka końcowych wyników prac w ramach projektu dotyczących analizy genomicznej. Obejmują one stacjonarne i wieloplatformowe oprogramowanie open source UGENE, które obejmuje pakiet powszechnie stosowanych narzędzi bioinformatycznych. Konsorcjum VIROGENESIS współpracowało też z firmą LUCIAD, która opracowuje oprogramowanie na potrzeby geoprzestrzennych rozwiązań związanych ze świadomością sytuacyjną. Współpraca ta służy przygotowaniu platformy dla dynamicznych wizualizacji oraz identyfikacji czynników ekologicznych, demograficznych i epidemiologicznych, które wpływają na rozprzestrzenianie się wirusów. Wykorzystanie aplikacji VIROGENESIS w przypadku obecnych wybuchów epidemii wirusowych W trakcie ostatniej epidemii Eboli zaangażowano kilka międzynarodowych zespołów badawczych do sekwencjonowania wirusa Ebola na potrzeby badań i działań związanych ze zdrowiem publicznym. Oprogramowanie VIROGENESIS okazało się być bardzo przydatne w kilku badaniach nad niedawnymi wybuchami epidemii w Afryce i Ameryce Południowej. Sieć badawcza Genome Detective identyfikuje patogeny wirusowe bezpośrednio na podstawie danych NGS. Wykorzystując te dane oraz dodatkowe narzędzia VIROGENESIS, można stosować zawierające wiele informacji wizualizacje i dynamiczne graficzne interfejsy użytkowników, by monitorować epidemie w czasie rzeczywistym i przestrzeni. W badaniu przedstawiono przykład wydajnego wykrywania wirusowych patogenów. Badacze wykorzystali narzędzia VIROGENESIS oraz dane z sekwencjonowania w czasie rzeczywistym na potrzeby genomicznego, przestrzennego i epidemiologicznego nadzoru nad epidemią wirusa żółtej gorączki w Brazylii w 2018 r. VIROGENESIS umożliwia skuteczną analizę danych NGS na potrzeby wykrywania wirusów, diagnostyki i monitorowania epidemii. Według koordynatorów projektu: „Wraz ze zwiększonym zakresem pozyskiwania genomicznych danych NGS z epidemii wirusowych oraz wybuchów epidemii przewidujemy zwiększony zakres zastosowania narzędzi VIROGENESIS na potrzeby analizy takich danych w sytuacjach, w których konieczne jest bardzo szybkie rozpoznanie typu wirusa i źródła zakażenia”.
Słowa kluczowe
VIROGENESIS, NGS, bioinformatyka, oprogramowanie, wybuchy epidemii wirusowych, wykrywanie wirusów