Bakterielle Genomanalysen für bahnbrechenden Nachweis von Antibiotikaresistenzen innerhalb eines Tages
Wie alle Organismen müssen sich auch Bakterien ständig veränderten Umweltbedingungen anpassen, u. a. Temperaturveränderungen, Nährstoffmangel und – was besonders kritisch ist – antibiotischen Wirkstoffen. Mutationen bzw. Veränderungen des DNA-Codes sind die wichtigsten Werkzeuge bei diesem evolutionären Prozess, sodass schon kleinste Veränderungen einzelner Nukleotide Erfolg haben können.
Antibiotika-Krise: gefährliche Entstehung von Antibiotikaresistenzen
Bakterien, die vor 50 Jahren noch auf Antibiotika ansprachen, passten ihre genetischen Mechanismen inzwischen so an, dass die Wirkung von Antibiotika gegen tödliche Infektionserreger nicht mehr gewährleistet ist. Ein alarmierender UN-Bericht zum Thema Antibiotikaresistenzen warnt ausdrücklich vor dem Risiko, in zunehmenden Maße häufige Infektionserkrankungen nicht mehr behandeln zu können. Ohne wirksame Gegenmaßnahmen könnten resistenten Infektionserregern bis 2050 weltweit jährlich 10 Millionen Menschen zum Opfer fallen. Bakterielle Evolution findet seit Milliarden von Jahren statt. Das Ziel ist, sich immer besser an die Umgebung anzupassen. Wenig überraschend gelingt es Bakterien so auch immer besser, Resistenzen gegen Antibiotika zu entwickeln. „Anders als nicht resistenten Bakterien gelingt es resistenten Erregern besser, ihre Virulenz zu erhalten und sich auch nach einem Antibiotikaangriff weiter zu vermehren“, erläutert Csaba Pál, Koordinator des EU-finanzierten Projekts Aware. „Trotz weltweiter Dringlichkeit ziehen sich große Pharmaunternehmen aus der Forschung an neuen Antibiotika zurück, was eine durchaus beängstigende Entwicklung ist“, fügt Pál hinzu. Pál führt hier die Millionenverluste von Pharmaunternehmen an, wenn sich z. B. erst in der klinischen Spätphase herausstellt, dass sich Resistenzen bilden können.
Screening evolutionärer Veränderungen in kürzerer Zeit
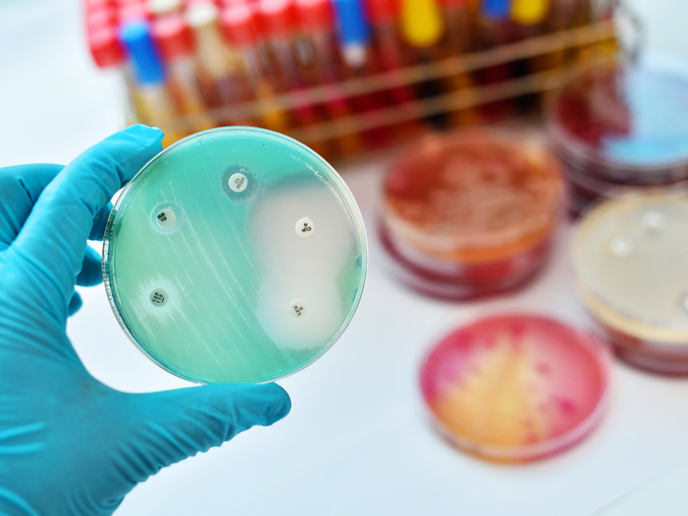
Angesichts des zunehmenden Investitionsrisikos entwickelte die Arbeitsgruppe um Pál eine bahnbrechende Methode, um bei neuen Antibiotikakandidaten in sehr frühen Phasen der klinischen Entwicklung das Risiko einer Resistenzbildung auszuschließen. „Mit unserem genomischen Verfahren DIvERGE kann eine ganze Molekülbibliothek auf einmal untersucht werden, um innerhalb weniger Tage Resistenzen aufzuzeigen. DIvERGE (directed evolution with random genomic mutations) bedeutet ‚gerichtete Evolution mit zufälligen genomischen Mutationen‘ und erhöht die Mutationsrate entlang bestimmter Regionen im Bakteriengenom auf das Millionenfache“, wie er erläutert. „In der Regel insertieren wir Mutationen an fünf Loci des Bakteriengenoms, wo mutmaßliche Resistenzen entstehen können. Labortechnisch können damit in nur ein oder zwei Tagen Milliarden Mutationskombinationen erzeugt werden.“
Machbarkeitstests
Diese hochspezifische und kontrollierte Anregung bakterieller Mutagenese könnte eine wirksame Strategie für die Wirkstoffforschung sein. Die Frage für Pál war jedoch: „Ist diese Methode lediglich eine fixe Idee, oder könnte sie sich tatsächlich in der Praxis bewähren?“ Für ihre Machbarkeitsstudie testeten Pál und sein Team Gepotidacin, ein potenzielles Erstlinien-Antibiotikum, das sich derzeit in klinischen Phase-III-Studien befindet. Bereits nach zwei Tagen enthüllte DIvERGE zwei spezifische Mutationen, die das Antibiotikum gänzlich wirkungslos machten. „Eine der Mutationen wurde sogar schon in vielen pathogenen Bakterien nachgewiesen, sodass viele dieser Bakterien nur noch eine Mutation oder Entwicklungsstufe vor der Resistenzbildung gegen Gepotidacin stehen“, erklärt Pál. Für die künftige Markteinführung der Technologie schlossen die Forschenden einen nicht-exklusiven Lizenzvertrag mit einem in Israel ansässigen Unternehmen, das Phagenwirkstoffe entwickelt. Die Arbeitsgruppe ist überzeugt davon, dass sich mit DIvERGE Resistenzprofile dieser Viren erstellen lassen, um sie als vielversprechende Alternativen zu Antibiotika gegen pathogene bakterielle Erreger einzusetzen. Die Angst vor schneller Resistenzbildung ist eine der größten Hürden in der Arzneimittelentwicklung. „Wird dieses Risiko in der Frühphase der Arzneimittelentwicklung entdeckt, könnten Pharmaunternehmen bei der Auswahl aussichtsreicher Wirkstoffkandidaten unterstützt werden, sodass keine Milliardeninvestitionen für neue Medikamente verschwendet werden, die bald sowieso nicht mehr wirken“, schließt Pál.
Schlüsselbegriffe
Aware, Resistenz, Bakterien, Antibiotika, Mutation, Pharmaunternehmen, Genom, DIvERGE, gerichtete Evolution