Wie Körper Infektionen bekämpft, gibt Hinweise auf Magenkrebsrisiko
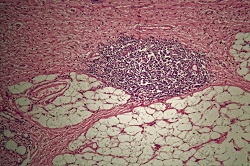
Die am HELICOMARK-Projekt arbeitenden, von der EU finanzierten Forscher haben Wege gefunden, um das Magenkrebsrisiko anhand von Blutproben zu bewerten. Dabei diente die körpereigene Immunantwort als Werkzeug. Weltweit sterben jährlich ungefähr 700 000 Menschen an Magenkrebs, der dritthäufigsten Krebstodesursache. Insgesamt sind in Japan, Korea und China fast 70 % aller Fälle zu finden, aber auch in Lateinamerika und Osteuropa ist die Krankheit häufig anzutreffen, und die Patientinnen und Patienten haben im Allgemeinen eine schlechte Überlebensprognose. Hauptursache für Magenkrebs ist der Keim Helicobacter pylori. „H. pylori als Bakterium ist genetisch sehr variabel und die Variation folgt mehr oder weniger der ethnischen Zugehörigkeit, wobei ostasiatische Versionen von H. pylori kanzerogener sind“, sagt Projektkoordinator Professor Samuel Lundin von der Abteilung für Mikrobiologie und Immunologie an der Universität Göteborg, Schweden. „Wir haben die Antikörperreaktionen auf H. pylori genauestens analysiert, um die in den Blutproben vorhandenen risikoreicheren H. pylori-Bakterien zu lokalisieren, und zu untersuchen, wie dies mit dem Magenkrebsrisiko in Verbindung zu bringen ist.“ Peptid-Arrays Die Forscher erstellen Peptid-Arrays, um die Immunantwort eines Patienten auf H. pylori durch Synthetisieren von Peptiden auf einem Glaschip zu beschreiben. „Es stehen uns Technologien zur Verfügung, mit denen wir eine Aminosäure hinzufügen, diese reagieren lassen, sie dann wegwaschen und eine weitere Aminosäure hinzufügen können, bis eine Peptidsequenz auf dem Glaschip entsteht“, erläutert Professor Lundin. Man fügte den 200.000 Peptiden auf dem Chip Serumproben aus Nikaragua, wo ein hohes Magenkrebsrisiko besteht, und Schweden hinzu. „In Abhängigkeit von den produzierten Antikörpern werden einige von ihnen an einige der Peptide binden. Dann fügt man einen weiteren Antikörper hinzu, der fluoreszenzmarkiert ist, damit diese Peptide aufleuchten”, erklärt Professor Lundin. „Der Assay belegt die Existenz dieser Antikörper, und so maskieren wir die Antikörper mit Hilfe des Peptidchips“, erklärt er. „Daraus erhalten wir einen Index, wie gut die Antikörper an die Peptide sowie an welche der 200.000 Peptide gebunden sind.” Auf diese Weise entsteht ein spezifischer „Fingerabdruck“ für jede Person, der dann mit denen verglichen werden kann, die den Krebs entwickelt haben. Bestandsaufnahme der Proteine „Wir haben alle Proteine zugeordnet, die bei Vorhandensein von H. pylori eine Antikörperreaktion auslösen, sowie sämtliche Epitope dieser Proteine (jene Teile der Proteine, an die sich Antikörper binden)“, sagt Professor Lundin „Wir haben nachgewiesen, dass nur zwei oder drei dieser Epitope bei den Patienten diagnostische Aussagen zulassen, und die anderen auch dann Antikörper produzieren, wenn der Patient keinen H. pylori hat.“ Dieses Verfahren zu Epitop-Mapping wurde patentiert. „Wir konnten die Peptide ermitteln, von denen wir wissen, dass ein hohes Magenkrebsrisiko besteht, wenn der Patient Antikörper gegen sie hat“, erklärt Professor Lundin. Der Schlüssel zum Erfolg des Projekts war der Zugang zu Blutproben sowie auch Biopsien von derselben Person. „Wir können die Bakterien aus den Biopsien kultivieren und die H. pylori-Stämme der Person mit Hilfe einer Genomanalyse untersuchen“, fährt er fort. Man analysiert dann die Antikörperantwort derselben Person, um jene Peptidsequenzen zu sehen, auf die sie reagieren. „Es ist unüblich, dass all diese Informationen zusammen vorliegen, und genau das war für uns ausschlaggebend.“ Professor Lundin erhielt ein Marie-Curie-Dreijahresstipendium, mit dem er zwei Jahre in Perth, Australien, finanziert, wo er mit dem Nobelpreisträger Barry Marshall, einem der beiden Entdecker von H. pylori, zusammenarbeitet.
Schlüsselbegriffe
HELICOMARK, Krebs, Immunologie, Biomarker, Fluoreszenz, Magenkrebs