Kontrolle der Genexpression über komplexe Netzwerke
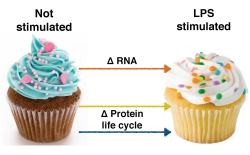
Lipopolysaccharide (LPS) auf der Oberfläche von Mikroben lösen sehr starke Immunreaktionen aus, indem sie mit Immunzellen wie Antigen-präsentierenden Zellen interagieren, insbesondere dendritischen Zellen (DC). Die Forschergruppe von EXPRESSION DYNAMICS untersuchte an DCs von Mausmodellen, wie Immunreaktionen gesteuert werden, die durch LPS auf bakteriellen Krankheitserregern ausgelöst werden. Analysen der RNA-Konzentration und Dynamik des Proteinzyklus (Produktion und Abbau) lieferten umfassenden Aufschluss darüber, wie sich die Genexpression bei dieser Immunreaktion verändert. So erklärt Marko Jovanovic, Forschungsleiter des Projekts: "Wir wollten ein globales Modell entwickeln, das zeigt, inwieweit jede regulatorische Schicht die jeweilige Genexpression beeinflusst." Ein neues Modell – das so genannte Cupcake Das Ergebnis ist ein Modell, bei dem neue zelluläre Funktionen – als Voraussetzung für eine schnelle Immunreaktion - primär durch Veränderung der RNA-Expression gesteuert werden. Zudem wird das bisherige Proteom so modifiziert, dass es sich an den aktivierten Zellzustand anpassen kann. Reguliert wird dies über den Proteinzyklus, also Produktion und/oder Abbau eines Proteins. "Wir nannten das Modell Cupcake, weil die Glasur zuerst ins Auge sticht. Veränderungen dieser oberen Schicht entsprechen dann den Modifizierungen, die durch die mRNA-Regulierung stattfinden. Der Kuchenteil selbst ist auch wichtig, wird aber oft und zu Unrecht vernachlässigt. Er entspricht den Housekeeping-Genen, die sich auch anpassen müssen. Unseren Ergebnissen zufolge kommt hier der Proteinzyklus ins Spiel", wie Marko Jovanovic weiter ausführt. Indem sie das systembiologische Modell auf die Stimulierung von Nervenzellen erweiterten, können die Forscher nun untersuchen, wie verschiedene Klassen von Genen räumlich und zeitlich in Zellkörpern und Dendriten dieser stimulierten Neuronen reguliert werden. Mit dem gleichen Proteomik- und Transkriptomikansatz wie für die LPS-stimulierten DCs sollen Tausende von Genen parallel sequenziert werden, um herauszufinden, ob Proteinveränderungen in bestimmten Zellkompartimenten (Zellkörpern und Dendriten) differenziell reguliert werden. Enthüllung neuer regulierender Gene Bislang wurde mittels Gen-Knockout nach den wichtigsten Regulatoren interessierender biologischer Prozesse gesucht. Dies ist für einfache Modellorganismen wie Hefe seit Jahrzehnten Gang und Gäbe, bei Säugetiersystemen, insbesondere Zelllinien, ist es aber schwieriger, da es oft um starke phänotypische Effekte wie Überleben geht. Im Gegensatz dazu entwickelte EXPRESSION DYNAMICS ein neues markerbasiertes Genom-Screening für Regulatoren, die die Expression interessierender Gene verändern. Mit diesem subtileren Ansatz lässt sich fast jeder biologische Prozess untersuchen. Mit dem modifizierten genomweiten CRISPR-Screen, bei dem ebenfalls DCs durch LPS stimuliert werden, entdeckten die Forscher viele unbekannte Regulatoren der LPS-induzierten Immunantwort. Dann wurden die Gene in funktionelle Module eingeteilt, je nach ihrer Reaktion auf die LPS-Stimulation. Genkontrolle für künftige klinische Anwendungen Die Patentanmeldung, die für die Bereitstellung, Nutzung und therapeutische Anwendung der CRISPR-CAS-Systeme und Modelle eingereicht wurde, zeigt im Ansatz den enormen Umfang dieser Technologie. Am wichtigsten ist dabei die Suche nach Genen, die an Leukozytenreaktionen für die Immunabwehr beteiligt sind, aber auch Modelle für gestörte Leukozytenaktivität und daraus resultierende Krankheiten. Der nächste Schritt, also die Prüfung möglicher chemischer und/oder Gentherapien, könnte von Bedeutung für künftige Therapien sein. Marko Jovanovic fasst dieses enorme biomedizinische Potenzial wie folgt zusammen: "Sobald man einen nachweisbaren Marker hat, kann mit diesem markerbasierten genomweiten Screen in primären Säugetierzellen für fast jedes System ein ergebnisoffenes genetisches Screening durchgeführt werden, um primäre therapeutische Zielstrukturen zu finden."
Schlüsselbegriffe
Kontrolle der Genexpression, Immunantwort, EXPRESSION DYNAMICS, dendritische Zelle, Nervenzelle