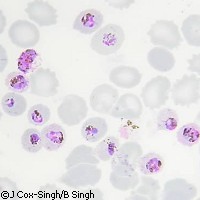
Naukowcy sekwencjonują genomy dwóch drobnoustrojów malarii
Dwa międzynarodowe zespoły naukowców wykonały sekwencjonownie genomów dwóch najważniejszych drobnoustrojów wywołujących malarię: Plasmodium vivax oraz Plasmodium knowlesi. Wyniki uwypuklają podobieństwa i różnice pomiędzy dwoma gatunkami Plasmodium, które wywołują malarię, dzięki czemu być może uda się opracować nowe leki i szczepionki do zwalczania tej śmiertelnej choroby. Malarię wywołują drobnoustroje z rodzaju Plasmodium, które przenoszone są pomiędzy ludźmi przez pewne gatunki komarów. Znanych jest co najmniej pięć odmian Plasmodium, które powodują malarię u ludzi: P. falciparum, P. vivax, P. ovale, P. malariae i P. knowlesi. Około trzy-czwarte przypadków zakażeń i 90% zgonów chorych na malarię wywoływanych jest przez P. falciparum, którego genom udało się odczytać w 2002 r. W trakcie najnowszych badań, o których donosi czasopismo Nature, naukowcy rozwikłali genom P. vivax, drugiego najbardziej śmiercionośnego szczepu chorobotwórczego, oraz P. knowlesi, który coraz częściej rozpoznawany jest jako główna przyczyna malarii u ludzi, szczególnie w południowo-wschodniej Azji. P. vivax jest główną przyczyną malarii poza Afryką i około 2,6 mld ludzi jest zagrożonych chorobą z jego powodu, głównie w Azji i Ameryce Łacińskiej. Inaczej niż P. falciparum, P. vivax rozwija się w bardziej umiarkowanych klimatach i w trakcie chłodniejszych pór roku. Choć rzadko śmiertelne, zakażenie P. vivax charakteryzuje się nawrotami choroby, co może ciągnąć się miesiącami. Powszechna odporność na leki powoduje, że coraz trudniej leczyć malarię wywołaną bakterią P. vivax. Analiza genomu P. vivax odkryła rodziny genów, które pomagają drobnoustrojowi włamać się do krwinek wcześniej nieznanymi kanałami. Odkryto także geny utajone - tę informację można by wykorzystać do opracowania leków, których zadaniem będzie zakłócić stan uśpienia drobnoustroju. "Nawroty malarii wywołanej przez Plasmodium vivax są poważnym wyzwaniem tak dla naukowców, jak i dla lekarzy" - powiedział Anthony Fauci z Państwowego Instytutu Alergii i Chorób Zakaźnych w USA. "Uzyskanie kompletnego genomu P. vivax daje nadzieje na nowe perspektywy w badaniach biologicznych nad wywoływaną przez ten drobnoustrój malarią oraz nowe możliwości w terapii i szczepieniach." W innym artykule, który pojawił się w Nature, opisano genom P. knowlesi. Naturalnym żywicielem szczepu jest makak jawajski, ale coraz częściej znajduje się go u ludzi, szczególnie w południowo-wschodniej Azji. Obecnie wiele przypadków wcześniej łączonych z P. malariae przypisuje się P. knowlesi. Trudno jest odróżnić te dwa szczepy używając tylko mikroskopu. Podobnie jak w przypadku P. vivax, opisanie genomu P. knowlesi przyniosło kilka niespodzianek. Na przykład posiada on geny, które wydają się podszywać pod ludzkie geny biorące udział w regulacji układu odpornościowego. Naukowcy podejrzewają, że te pasożytnicze geny pomagają P. knowlesi wpływać na zdolność rozpoznania zainfekowanych krwinek przez ludzkiego żywiciela. "Nasze badania dowodzą wagi sekwencjonowania innych genomów malarii w rozwikłaniu nadal nierozpoznanych i fascynujących aspektów biologicznych drobnoustrojów malarii" - komentuje dr Arnab Pain z Wellcome Trust Sanger Institute, w którym także prowadzone są prace nad opisaniem genomów pozostałych dwóch gatunków Plasmodium, o których wiadomo, że są zakaźne dla ludzi. "Zazwyczaj decydujące geny, które - jak podejrzewamy - pomagają drobnoustrojowi uniknąć wykrycia i zniszczenia przez system obronny żywiciela, porozrzucane są po całym genomie. W innych gatunkach, które przebadaliśmy, geny te najczęściej lokują się na czubkach chromosomów." "Jest to pierwszy opis genomu drobnoustroju malarii małpiej. Jest on intrygujący i zadziwiający, ale także daje nam nowe możliwości w walce z malarią" - dodaje dr Alan Thomas z Centrum Badań Biomedycznych Nad Naczelnymi w Rijswijk, Holandia. "P. knowlesi jest blisko spokrewniony z drugą najczęstszą przyczyną malarii u ludzi - P. vivax. Dzięki zrozumieniu genetycznej architektury obydwu drobnoustrojów, będziemy w stanie skuteczniej wykorzystać wyniki badań nad P. knowlesi w analizach innych ludzkich drobnoustrojów. Co równie ważne, znajomość genomu pomoże zrozumieć przypadki malarii wywołanej przez P. knowlesi u ludzi." Wsparcie UE dla badań nad P. knowlesi pochodziło z projektu BioMalPar (Biologia i patologia drobnoustrojów malarii), który finansowany jest w ramach tematu "Nauki o życiu, genomika i biotechnologia na rzecz zdrowia" Szóstego Programu Ramowego (6PR) oraz projektu VIRMAL (Struktura, funkcje i potencjał szczepieniowy wielogenowej rodziny vir w malarii), finansowanego w ramach tematu "Jakość życia i zarządzanie żywymi zasobami" Piątego Programu Ramowego (5PR).